Sektion Klinische Virusgenomik
Arbeitsgebiete
Die Sektion Klinische Virusgenomik unter der Leitung von wurde im Januar 2021 gegründet und beschäftigt sich mit der molekularen Epidemiologie von Viren und deren Evolution auf Wirtsebene. Hierzu verwenden wir u. a. neueste Next Generation-Sequencing (NGS) Technologien.
Wir stehen in enger Kooperation mit den weiteren virologischen Arbeitsgruppen im Institut, um die Virusevolution sowohl im infizierten Wirt als auch in mittels Zellkultur isolierten Viren eingehender zu analysieren. So konnten wir zeigen, dass bei SARS-CoV-2 Langzeit-infizierten, immunsupprimierten Patienten Varianten selektiert werden, die möglichweise immun- oder Therapie-evasiv sind (siehe unter Ausgewählte Publikationen). Dadurch könnte eine Evolution des Virus innerhalb eines immun-kompromittierten Patienten zu Virus-Varianten führen, die besser mit dem Immunsystem des Wirts oder medikamentöser Intervention zurechtkommen und die Grundlage für Virus-Varianten mit einem erhöhten pandemischen Potential bilden.
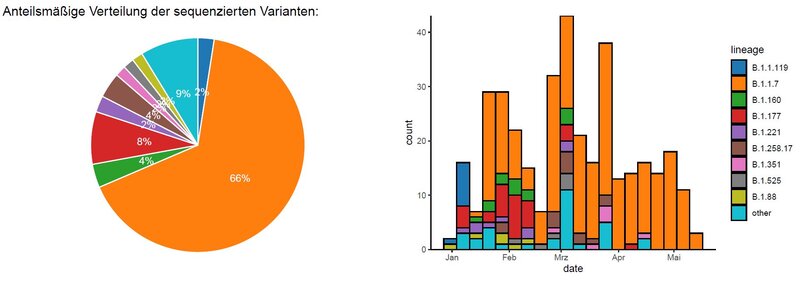
Am Beispiel von SARS-CoV-2 analysieren wir die aktuelle Dynamik des SARS-CoV-2 Infektionsgeschehen und das Auftreten von neuen Virus-Varianten. Hierzu engagieren wir uns im Rahmen des Netzwerks universitäre Medizin (NUM) und in enger Kooperation mit dem Institut für Infektionsprävention bei der molekularen Surveillance von SARS-CoV-2. Weiterhin untersuchen wir mit Hilfe von NGS Transmissionsketten im Krankenhaus, um diese besser zu verstehen. Dies soll dazu führen, Übertragungsketten frühzeitiger zu erkennen und Risikofaktoren für nosokomiale Übertragungen zu definieren.
Unsere Erfahrungen aus der Pandemie übertragen wir aktuell auf weitere virale Erreger. Vor allem fokusieren wir uns darauf, Amplikon-basierte Vollgenomsequenzierungen für hochdiverse Viren zu etablieren. Hierfür verwenden ein eigens entwickeltes Softwarepaket (https://github.com/jonas-fuchs/varVAMP), um Primer für diese Technologie in silico zu designen. In diesem Projekt kooperieren wir unter anderem mit Mathias Schemmerer und Jürgen Wenzel von der Universität Regensburg (für Hepatitis E) und Julian Kreiblich und Sindy Böttcher vom Robert Koch Institut (für Poliovirus). Durch diese neu entwickelte Methoden können virale Erreger einfach und kostengünstig direkt aus diagnostischen Proben sequenziert werden. Perspektivisch sollen diese Arbeiten auch auf weitere krankenhausrelevante Virusinfektionen ausgedehnt werden.
Im Rahmen eines naturwissenschaftlichen Promotionsvorhabens (Frau L. Jaki) werden zurzeit Fragen bei der Resistenzentstehung von HSV-1 und HSV-2 Infektionen in Kooperation mit dem Konsiliarlabor für Herpes-simplex-Virus 1/2 und Varizella-Zoster-Virus sowie der AG Ruzsics am Institut bearbeitet.
In Kooperation mit der ESCMID Study Group Genomic & Molecular Diagnostics (Prof. G. Greub) führen wir zurzeit einen europäischen Pilot-Ringversuch zur NGS-Diagnostik von Mpox/Affenpocken durch.
Mitarbeitende
- Dr. Jonas Fuchs, Postdoc
- Dr. Zsolt Ruzsics (see Research Teams, AG Ruzsics)
- Lena Jaki, PhD Student
- Anastasija Maks, Technician
- Johanna Kleine, MD Student
Kooperationen
- Prof. Hajo Grundmann, Dr. Sandra Reuter und Dr. Stefanie Kramme, Institut für Infektionsprävention und Krankenhaushygiene, Freiburg
- Prof. Hartmut Hengel, Konsiliarlabor für Herpes-simplex-Virus 1/2 und Varizella-Zoster-Virus, und Dr. Zsolt Ruzsics, Institut für Virologie, Freiburg
- Prof. Martin Schwemmle und Prof. Georg Kochs, Institut für Virologie, Freiburg
- PD Dr. Roland Elling, Pädiatrische Infektiologie, Freiburg
- Dr. Wolfgang Maier und Dr. Björn Grüning, Galaxy Europe, Standort Freiburg
- Prof. Adrian Egli, Klinische Mikrobiologie, Unispital Basel, Schweiz
- Prof. Gilbert Greub, Mikrobiologie, Universitätsspital Lausanne, Schweiz
- Dr. Sindy Böttcher und Dr. Sandra Niendorf, RKI, Berlin
- Prof. Jürgen Wenzel und Dr. Mathias Schemmerer, Institut für Mikrobiologie, Regensburg
Ausgewählte Publikationen
Total escape of SARS-CoV-2 from dual monoclonal antibody therapy in an immunocompromised patient.
Jaki L, Weigang S, Kern L, Kramme S, Wrobel AG, Grawitz AB, Nawrath P, Martin SR, Dähne T, Beer J, Disch M, Kolb P, Gutbrod L, Reuter S, Warnatz K, Schwemmle M, Gamblin SJ, Neumann-Haefelin E, Schnepf D, Welte T, Kochs G, Huzly D, Panning M, Fuchs J. Nat Commun. 2023 Apr 10;14(1):1999. doi: 10.1038/s41467-023-37591-w. PMID: 37037847
Hornuss D, Daehne T, Goetz V, Mueller M, Usadel S, Lorz A, Mockenhaupt M, Huzly D, Bierbaum S, Fuchs J, Jaki L, Falcone V, Kochs G, Panning M, Rieg S. Clin Microbiol Infect. 2023 Jan;29(1):112.e5-112.e9. doi: 10.1016/j.cmi.2022.09.012. Epub 2022 Sep 23. PMID: 36155255
Hirsiger JR, Weigang S, Walz AC, Fuchs J, Daly ML, Eggimann S, Hausmann O, Schwemmle M, Kochs G, Panning M, Warnatz K, Recher M, Berger CT. J Allergy Clin Immunol Pract. 2022 Sep;10(9):2452-2455.e3. doi: 10.1016/j.jaip.2022.06.020. Epub 2022 Jun 29. PMID: 35779780
Antibody escape and global spread of SARS-CoV-2 lineage A.27.
Kaleta T, Kern L, Hong SL, Hölzer M, Kochs G, Beer J, Schnepf D, Schwemmle M, Bollen N, Kolb P, Huber M, Ulferts S, Weigang S, Dudas G, Wittig A, Jaki L, Padane A, Lagare A, Salou M, Ozer EA, Nnaemeka N, Odoom JK, Rutayisire R, Benkahla A, Akoua-Koffi C, Ouedraogo AS, Simon-Lorière E, Enouf V, Kröger S, Calvignac-Spencer S, Baele G, Panning M, Fuchs J. Nat Commun. 2022 Mar 3;13(1):1152. doi: 10.1038/s41467-022-28766-y. PMID: 35241661
Wegner F, Roloff T, Huber M, Cordey S, Ramette A, Gerth Y, Bertelli C, Stange M, Seth-Smith HMB, Mari A, Leuzinger K, Cerutti L, Harshman K, Xenarios I, Le Mercier P, Bittel P, Neuenschwander S, Opota O, Fuchs J, Panning M, Michel C, Hallin M, Demuyser T, De Mendonca R, Savelkoul P, Dingemans J, van der Veer B, Boers SA, Claas ECJ, Coolen JPM, Melchers WJG, Gunell M, Kallonen T, Vuorinen T, Hakanen AJ, Bernhoff E, Hetland MAK, Golan Berman H, Adar S, Moran-Gilad J, Wolf DG, Leib SL, Nolte O, Kaiser L, Schmutz S, Kufner V, Zaheri M, Trkola A, Aamot HV, Hirsch HH, Greub G, Egli A. J Clin Microbiol. 2022 Jan 19;60(1):e0169821. doi: 10.1128/JCM.01698-21. Epub 2021 Nov 10. PMID: 34757834
Weigang S, Fuchs J, Zimmer G, Schnepf D, Kern L, Beer J, Luxenburger H, Ankerhold J, Falcone V, Kemming J, Hofmann M, Thimme R, Neumann-Haefelin C, Ulferts S, Grosse R, Hornuss D, Tanriver Y, Rieg S, Wagner D, Huzly D, Schwemmle M, Panning M, Kochs G. Nat Commun. 2021 Nov 4;12(1):6405. doi: 10.1038/s41467-021-26602-3. PMID: 34737266

Ärztlicher Direktor:
Prof. Dr. med. Hartmut Hengel
hartmut.hengel@uniklinik-freiburg.de
| Sekretariat: Kristina Gendrisch Telefon: 0761 270-83480 Telefax: 0761 270-83479 E-Mail: kristina.gendrisch@uniklinik-freiburg.de | Administration:
| Pforte: Jutta Schneeberger Telefon: 0761 270-83700 Telefax: 0761 270-83703 E-Mail: jutta.schneeberger@uniklinik-freiburg.de | |
| Probeneingangslabor: | Abrechnungsbüro: | ||
| Probeneingangserfassung, allgemeine Einsenderberatung, Probenverarbeitung und –archivierung Tel.: 0761 - 270-83460 Fax: 0761 - 270-83449 E-Mail: diagnostik.virologie@uniklinik-freiburg.de | Bereich: Privat-, BG-Abrechnung, PVS, interne Leistungsabrechnung Telefon: 0761 270-83458 Fax: 0761 270-83449 E-Mail: sabine.ay@uniklinik-freiburg.de, anna.kosog@uniklinik-freiburg.de | Bereich: Externe Krankenhäuser, Konsiliarlabor, Selbstzahler, Ämter, KV- Ermächtigung Telefon: 0761 270-83457 Fax: 0761 - 270-83449 E-Mail: martina.deutschmann@uniklinik-freiburg.de | |