IIK Arbeitsgruppe Mikrobielle Genomanalyse
- Ganzgenomsequenzierung krankenhausrelevanter Pathogene -Über uns
Wir benutzen Gesamtgenomsequenzierung als Tool, um uns anzuschauen, was ein Bakterium ausmacht. Indem wir seinen genetischen Kode auslesen, haben wir die größtmögliche Auflösung, um uns über mögliche Übertragungen auf Krankenhaus-Stationen zu informieren, was uns ermöglicht, Ausbrüche zu untersuchen. Weiterhin können wir aus den generierten Daten auch über Antibiotika-Resistenzen lernen. Wir sind in einer Anzahl von Projekten mit internen und externen Kollaborateuren involviert. Eines unserer Hauptaugenmerke liegt auf der Ausbreitung von multiresistenten Gram-negativen Klonen in Krankenhausnetzwerken. Dabei arbeiten wir eng mit Wissenschaftlern am Wellcome Trust Sanger Institute (Cambridge, UK), Big Data Institute (Oxford, UK), University Medical Center Groningen (Groningen, Niederlande), Charité Berlin (Berlin, Deutschland) und Justus-Liebig-Universitätsklinik (Gießen, Deutschland) zusammen.
Zentrale Themen
- Ganzgenomsequenzierung zur Infektionsprävention und -kontrolle
- Verständnis der Übertragungsdynamiken und des Einflusses der Krankenhausumgebung auf die Transmission
- Entwicklung von Sequenzierungs- und Analyseverfahren für neue Herausforderungen: Plasmidübertragung, metagenomische Diagnostik
Ausgewählte aktuelle Publikationen
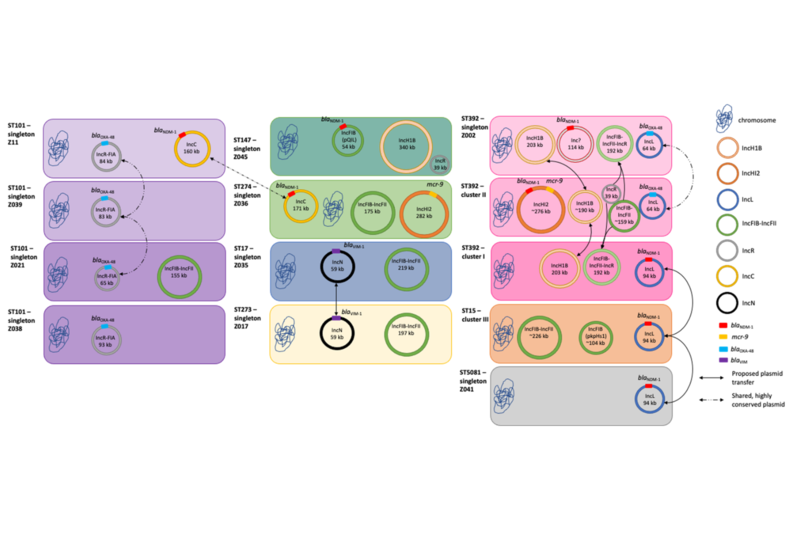
- Akintayo I, et al. Tracking clonal and plasmid transmission in colistin- and carbapenem-resistant Klebsiella pneumoniae. mSystems 2025 10(2):e01128-24.
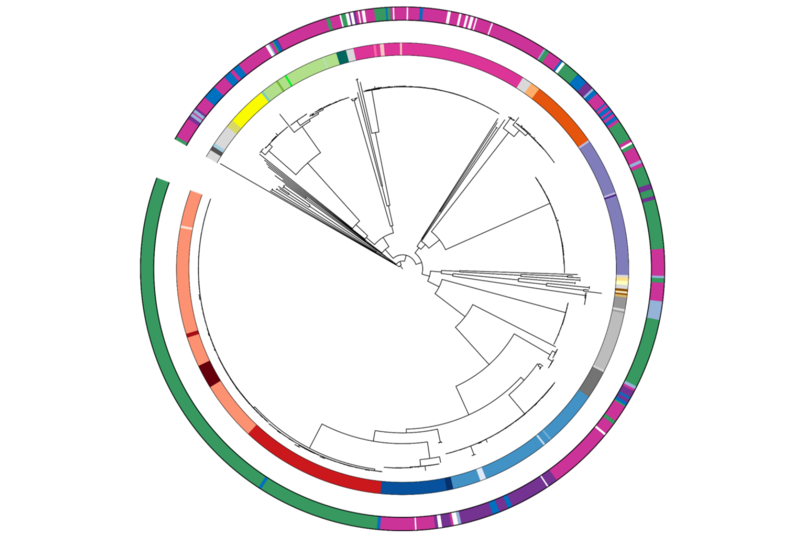
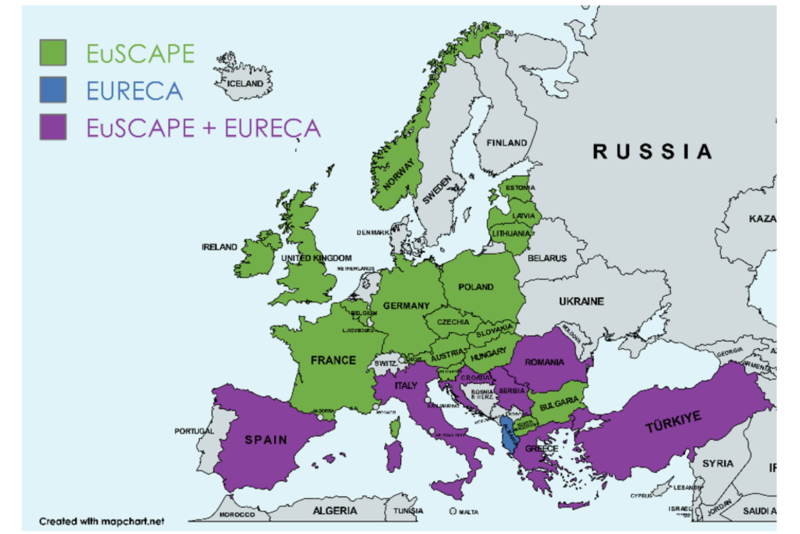
- Budia-Silva M, et al. International and regional spread of carbapenem-resistant Klebsiella pneumoniae in Europe. Nature Communications 2024 15(1):5092.
- Reuter S, et al. A global view on carbapenem-resistant Acinetobacter baumannii. mBio 2023 14(6):e02260-23.
Technologie
- Illumina MiSeq & Oxford Nanopore Technology GridION
- Expertise in bakterieller Gesamtgenomsequenzierung & Analyse
- Bioinformatische Expertise
Gefördert durch

Dr. biol. Sandra Reuter
Tel.: +49 (0) 761 270 82350
Fax: +49 (0) 761 270 83030