Liquid Biopsy
Über unser Labor
Die moderne Onkologie strebt nach immer präziseren, gezielteren und personalisierten diagnostischen und therapeutischen Mitteln im Kampf gegen Krebserkrankungen. Der Nachweis von Biomarkern aus Körperflüssigkeiten (sog. Liquid Biopsy) spielt hierbei eine immer größere Rolle. Im engeren Sinne bezeichnet der Begriff Liquid Biopsy in diesem Zusammenhang den Nachweis von im Blut zirkulierender Tumor-DNA (ctDNA) / RNA oder zirkulierenden Tumorzellen.
Die Liquid Biopsy ist durch einige entscheidende Vorteile gekennzeichnet: Sie ist durch eine einfache Blutentnahme zu jedem beliebigen Zeitpunkt nicht-invasiv durchführbar. Deshalb kann sie eine Alternative zur konventionellen Gewebe-Biopsie sein, wenn diese aus verschiedenen Gründen nicht durchgeführt werden kann oder Tumormaterial nicht ausreichend vorhanden ist. Durch die hohe Spezifität und Sensitivität eignet sich die Liquid Biopsy hervorragend zur Bestimmung von Blut-Biomarkern zur Verlaufsbeobachtung von Krebserkrankungen. Außerdem bildet insbesondere ctDNA im Blut von Patienten alle Tumormanifestationen im Körper ab und kann daher zur Analyse der Tumor-Heterogenität herangezogen werden.
Im Liquid Biopsy Labor der Klinik für Innere Medizin I des Universitätsklinikums Freiburg entwickeln und optimieren wir PCR-basierte Methoden (insb. digital droplet PCR, ddPCR) und Next-Generation Sequencing- (NGS-)- Technologien für die translationale Biomarker-Forschung und bringen sie in der klinischen Routine zur Anwendung.
Leitung
KontaktDr. Julius Wehrle
Funktionsoberarzt
Kontakt:
Universitätsklinikum Freiburg
Klinik für Innere Medizin I – Liquid Biopsy Labor
Hugstetter Straße 55 | 79106 Freiburg
Ulrike Philipp oder Inga Gebhard
Telefon: +49 761 270 33850
ulrike.philipp@uniklinik-freiburg.de
inga.gebhard @uniklinik-freiburg.de
Allgemein erreichbare E-Mail-Adresse:
med1.liquidbiopsy-labor@uniklinik-freiburg.de
Liquid Biopsy
Verfahren in der KlinikDas Liquid Biopsy Labor entwickelt und etabliert PCR-basierte Testverfahren (digital droplet PCR, ddPCR) zur Untersuchung von Tumor-Mutationen im Blut von Krebspatienten. In der folgenden Übersicht finden Sie die bisher von uns entwickelten Assays, welche in Forschungsprojekten zur Beantwortung wissenschaftlicher Fragestellungen zum Einsatz kommen:
Leistungsverzeichnis vom Liquid Biopsy Labor
Stand: 05.03.2023
Drei dieser ddPCR-Assays können in unserem Labor angefordert werden:
EGFR T790M: Diese Mutation spielt bei Patienten mit fortgeschrittenem Nichtkleinzelligem Lungenkarzinom (NSCLC) und einer aktivierenden EGFR-Aberration, bei denen es unter Therapie mit einem EGFR Tyrosinkinase-Inhibitor (TKI) zu einem Progress und somit zu einer Resistenz gekommen ist, eine wichtige Rolle.
KRAS Kodon 12/13: Das KRAS-Gen gehört zu den häufig somatisch mutierten Genen in diversen humanen Tumoren (insbes. Kolonkarzinom, Pankreaskarzinom, Lungenkarzinom). Diese somatische Mutationen führen zu einer konstitutiven Aktivierung des KRAS-Proteins und damit des RAS/RAF/MEK/ERK-Signalweges unter Ausschluss einer Regulation durch EGFR.
BRAF V600E: Die BRAF V600E-Mutation führt zu einer Aktivierung des BRAF-Onkogens und damit zu unkontrolliertem Zellwachstum. Diese Mutation spielt eine wichtige Rolle in unterschiedlichen Krebsarten (insbes. Malignes Melanom, Kolonkarzinom, Schilddrüsenkarzinom, Haarzell-Leukämie oder Lungenkarzinom), wobei zielgerichtete Therapien verfügbar sind (sog. BRAF-Inhibitoren).
Im Folgenden finden Sie Informationen zur Anforderung eines Liquid Biopsy-Tests zur Analyse von EGFR T790M, KRAS Kodon 12/13 und BRAF V600E und eine Anleitung zur korrekten Entnahme von Blut und Verschickung des Materials an unser Labor:
Forschung
Die Forschungsaktivitäten des Liquid Biopsy Lab Freiburg umfassen sowohl die Entwicklung und Optimierung modernster Liquid Biopsy-Analyseverfahren (Next-Generation Sequencing [NGS], digital droplet PCR [ddPCR]) als auch deren Anwendung zur translationalen Erforschung verschiedener Krebsbiomarker.
Unsere Forschungsprojekte beinhalten die folgenden Tumor-Entitäten:
- Maligne Melanome
- Nicht-kleinzellige Lungenkarzinome (NSCLC)
- Kolonkarzinome
- Gastrointestinale Stromatumoren (GIST)
- Sarkome [Link Wehrle Lab]
- Primäre Lymphome des zentralen Nervensystems (PCNSL) Scherer Lab
- Diffuse großzellige B-Zell-Lymphome (DLBCL) Scherer Lab
- Glioblastome und Lower-grade Gliome (LGG) Scherer Lab
Kollaborationen intern:
- Prof. Dr. Dr. Melanie Börries, Insitut für Bioinformatik und System-Medizin
- Prof. Dr. Marco Prinz, Institut für Neuropathologie
- Prof. Dr. Silke Lassmann, Institut für Klinische Pathologie
- Prof. Dr. Dr. Meyer, Klinik für Nuklearmedizin
- Dr. Peter Bronsert, Institut für Klinische Pathologie
- Dr. Frank Meiß, Klinik für Dermatologie und Venerologie
- Dr. Elisabeth Schorb, Klinik für Innere Medizin I
- Dr. Dietmar Pfeifer, Klinik für Innere Medizin I
- Dr. Miguel Waterhouse, Klinik für Innere Medizin I
- Dr. Peter Reinacher, Abteilung Stereotaktische und Funktionelle Neurochirurgie
- Dr. Dieter Hendrik Heiland, Klinik für Neurochirurgie
- PD Dr. Simone Hettmer, Pädiatrische Hämatologie und Onkologie
- Dr. David Braig, Klinik für Plastische und Handchirurgie
Kollaborationen extern:
- Prof. Dr. Nikolas von Bubnoff, Universitätsklinikum Lübeck
- Dr. Ralph Fritsch, Universitätsspital Zürich
- Prof. Dr. Gerald Illerhaus, Klinikum Stuttgart
- PD Dr. Dr. Benjamin Kasenda, Klinikum Stuttgart
- Dr. Lorenz Thurner, Universitätsklinikum des Saarlandes, Homburg/Saar
- Ash A. Alizadeh MD/PhD, Stanford University
- David M. Kurtz MD/PhD, Stanford University
- Henning Stehr PhD, Stanford University

Mitarbeiter*innen (alphabetisch):
- Sabine Bleul, MTA
- Maximilian Deuter, cand. med.
- Inga Gebhard, MTA
- Dr. Thomas Meyer, Arzt
- Dr. Jan Mitschke, Bioinformatiker
- Ulrike Philipp, MTA
- Dr. Michael Rassner, Arzt
Frühere Leitung:
Prof. Dr. Nikolas von Bubnoff (Ärztlicher Direktor Universitätsklinikum Lübeck)
E-Mail: Nikolas.vonBubnoff@uksh.de
Alumni:
- Jan Braune, cand. med
- Felipa Merkel, cand. med.
- Martina Jolic, MTA
- Silvia Waldeck, PhD-Student
Publikationen
Dynamic Risk Profiling Using Serial Tumor Biomarkers for Personalized Outcome Prediction
Kurtz DM*, Esfahani MS*, Scherer F*, Soo J, Jin MC, Liu CL, Newman AM, Dührsen U, Hüttmann A, Casasnovas O, Westin JR, Ritgen M, Böttcher S, Langerak AW, Roschweski M, Wilson WH, Gaidano G, Rossi D, Bahlo J, Hallek M, Tibshirani R, Diehn M, Alizadeh AA
* Co-first Autor
In Fett ==> Labormitglieder | * ==> Co-Erstautoren
2021
- Enhanced detection of minimal residual disease by targeted sequencing of phased variants in circulating tumor DNA.
Kurtz DM, Soo J, Co Ting Keh L, Alig S, Chabon JJ, Sworder BJ, Schultz A, Jin MC, Scherer F, Garofalo A, Macaulay CW, Hamilton EG, Chen B, Olsen M, Schroers-Martin JG, Craig AFM, Moding EJ, Esfahani MS, Liu CL, Dührsen U, Hüttmann A, Casasnovas RO, Westin JR, Roschewski M, Wilson WH, Gaidano G, Rossi D, Diehn M, Alizadeh AA.
Nat Biotechnol. 2021 Jul 22. doi: 10.1038/s41587-021-00981-w. Online ahead of print. PMID: 34294911
- Short Diagnosis-to-Treatment Interval Is Associated With Higher Circulating Tumor DNA Levels in Diffuse Large B-Cell Lymphoma.
Alig S, Macaulay CW, Kurtz DM, Dührsen U, Hüttmann A, Schmitz C, Jin MC, Sworder BJ, Garofalo A, Shahrokh Esfahani M, Nabet BY, Soo J, Scherer F, Craig AFM, Casasnovas O, Westin JR, Gaidano G, Rossi D, Roschewski M, Wilson WH, Meignan M, Diehn M, Alizadeh AA.
J Clin Oncol. 2021 Apr 28:JCO2002573. doi: 10.1200/JCO.20.02573. Online ahead of print. PMID: 33909455
- Longitudinal analysis of cell-free mutated KRAS and CA 19-9 predicts survival following curative resection of pancreatic cancer.
Hussung S, Akhoundova D, Hipp J, Follo M, Klar RFU, Philipp U, Scherer F, von Bubnoff N, Duyster J, Boerries M, Wittel U, Fritsch RM.
BMC Cancer. 2021 Jan 11;21(1):49. doi: 10.1186/s12885-020-07736-x. PMID: 33430810 Free PMC article. Clinical Trial.
- Colon and liver tissue damage detection using methylated SESN3 and PTK2B genes in circulating cell-free DNA in patients with acute graft-versus-host disease.
Waterhouse M, Pennisi S, Pfeifer D, Deuter M, von Bubnoff N, Scherer F, Strüssmann T, Wehr C, Duyster J, Bertz H, Finke J, Duque-Afonso J.
Bone Marrow Transplant. 2021 Feb;56(2):327-333. doi: 10.1038/s41409-020-01090-z. Epub 2020 Oct 20. PMID: 33082554
2020
- Personalized Treatment Selection and Disease Monitoring Using Circulating Tumor DNA Profiling in Real-World Cancer Patient Management.
Wehrle J, Philipp U, Jolic M, Follo M, Hussung S, Waldeck S, Deuter M, Rassner M, Braune J, Rawluk J, Greil C, Waller CF, Becker H, Duque-Afonso J, Illert AL, Fritsch RM, Meiss F, Duyster J, von Bubnoff N, Scherer F.
Diagnostics (Basel). 2020 Aug 2;10(8):550. doi: 10.3390/diagnostics10080550. PMID: 32748806 Free PMC article.
- Development and Clinical Validation of Discriminatory Multitarget Digital Droplet PCR Assays for the Detection of Hot Spot KRAS and NRAS Mutations in Cell-Free DNA.
Hussung S, Follo M, Klar RFU, Michalczyk S, Fritsch K, Nollmann F, Hipp J, Duyster J, Scherer F, von Bubnoff N, Boerries M, Wittel U, Fritsch RM.
J Mol Diagn. 2020 Jul;22(7):943-956. doi: 10.1016/j.jmoldx.2020.04.206. Epub 2020 May 4. PMID: 32376474
- Capturing Tumor Heterogeneity and Clonal Evolution by Circulating Tumor DNA Profiling.
Scherer F.
Recent Results Cancer Res. 2020;215:213-230. doi: 10.1007/978-3-030-26439-0_11.
2019
- Dynamic Risk Profiling Using Serial Tumor Biomarkers for Personalized Outcome Prediction
CELL
Kurtz DM*, Esfahani MS*, Scherer F*, Soo J, Jin MC, Liu CL, Newman AM, Dührsen U, Hüttmann A, Casasnovas O, Westin JR, Ritgen M, Böttcher S, Langerak AW, Roschweski M, Wilson WH, Gaidano G, Rossi D, Bahlo J, Hallek M, Tibshirani R, Diehn M, Alizadeh AA
2019. doi:https://doi.org/10.1016/j.cell.2019.06.011 - Circulating cKIT and PDGFRA DNA indicates disease activity in Gastrointestinal Stromal Tumor (GIST) INTERNATIONAL JOURNAL OF CANCER
Jilg S, Rassner M, Maier J, Waldeck S, Kehl V, Follo M, Philipp U, Sauter A, Specht K, Mitschke J, Lange T, Bauer S, Jost PJ, Peschel C, Duyster J, Gaiser T, Hohenberger P, von Bubnoff N
2019. doi:10.1002/ijc.32282 - Capturing Tumor Heterogeneity and Clonal Evolution by Circulating Tumor DNA Profiling
RECENT RESULTS CANCER RES.
Scherer F
2020;215:213-230. doi: 10.1007/978-3-030-26439-0_11
2018
- Circulating Tumor DNA Measurements As Early Outcome Predictors in Diffuse Large B-Cell Lymphoma JOURNAL OF CLINICAL ONCOLOGY
Kurtz DM*, Scherer F*, Jin MC, Soo J, Craig AFM, Esfahani MS, Chabon JJ, Stehr H, Liu CL, Tibshirani R, Maeda LS, Gupta NK, Khodadoust MS, Advani RH, Levy R, Newman AM, Duehrsen U, Huttmann A, Meignan M, Casasnovas RO, Westin JR, Roschewski M, Wilson WH, Gaidano G, Rossi D, Diehn M, Alizadeh AA
2018. doi: 10.1200/JCO.2018.78.5246 - Extracellular vesicles or free circulating tumor DNA: where to search for BRAF and cKIT mutations? NANOMEDICINE
Klump J, Philipp U, Follo M, Eremin A, Lehmann H, Nestel S, von Bubnoff N, Nazarenko I
2018. doi:10.1016/j.nano.2017.12.009
2017
- High-throughput sequencing for noninvasive disease detection in hematologic malignancies
BLOOD
Scherer F*, Kurtz DM*, Diehn M, Alizadeh AA
2017. doi:10.1182/blood-2017-03-735639 - Early Detection of Molecular Residual Disease in Localized Lung Cancer by Circulating Tumor DNA Profiling
CANCER DISCOVERY
Chaudhuri AA, Chabon JJ, Lovejoy AF, Newman AM, Stehr H, Azad TD, Khodadoust MS, Esfahani MS, Liu CL, Zhou L, Scherer F, Kurtz DM, Say C, Carter JN, Merriott DJ, Dudley JC, Binkley MS, Modlin L, Padda SK, Gensheimer MF, West RB, Shrager JB, Neal JW, Wakelee HA, Loo BW Jr, Alizadeh AA, Diehn M
2017. doi: 10.1158/2159-8290.CD-17-0716 - A specific spectral signature of serum and plasma-derived extracellular vesicles for cancer screening
NANOMEDICINE
Krafft C, Wilhelm K, Eremin A, Nestel S, von Bubnoff N, Schultze-Seemann W, Popp J,
Nazarenko I
2017. doi:10.1016/j.nano.2016.11.016 - Liquid Biopsy: Approaches to Dynamic Genotyping in Cancer
ONCOLOGY RESEARCH AND TREATMENT
von Bubnoff N
2017. doi:10.1159/000478864
2016
- Distinct biological subtypes and patterns of genome evolution in lymphoma revealed by circulating tumor
DNA SCIENCE TRANSLATIONAL MEDICINE
Scherer F*, Kurtz DM*, Newman AM*, Stehr H, Craig AF, Esfahani MS, Lovejoy AF, Chabon JJ, Klass DM, Liu CL, Zhou L, Glover C, Visser BC, Poultsides GA, Advani RH, Maeda LS, Gupta NK, Levy R, Ohgami RS, Kunder CA, Diehn M, Alizadeh AA
2016. doi:10.1126/scitranslmed.aai8545 - Integrated digital error suppression for improved detection of circulating tumor DNA
NATURE BIOTECHNOLOGY
Newman AM*, Lovejoy AF*, Klass DM*, Kurtz DM, Chabon JJ, Scherer F, Stehr H, Liu CL, Bratman SV, Say C, Zhou L, Carter JN, West RB, Sledge GW, Shrager JB, Loo BW Jr, Neal JW, Wakelee HA, Diehn M, Alizadeh AA
2016. doi:10.1038/nbt.3520 - Circulating tumor DNA profiling reveals heterogeneity of EGFR inhibitor resistance mechanisms in lung cancer patients
NATURE COMMUNICATIONS
Chabon JJ*, Simmons AD*, Lovejoy AF, Esfahani MS, Newman AM, Haringsma HJ, Kurtz DM, Stehr H, Scherer F, Karlovich CA, Harding TC, Durkin KA, Otterson GA, Purcell WT, Camidge DR, Goldman JW, Sequist LV, Piotrowska Z, Wakelee HA, Neal JW, Alizadeh AA, Diehn M
2016. doi:10.1038/ncomms11815 - Low Input Whole-Exome Sequencing to Determine the Representation of the Tumor Exome in Circulating DNA of Non-Small Cell Lung Cancer Patients
PLOS ONE
Dietz S, Schirmer U, Merce C, von Bubnoff N, Dahl E, Meister M, Muley T,Thomas M, Sultmann H 2016. doi:10.1371/journal.pone.0161012 - Sensitive and accurate quentification of JAK2 V617F mutation in chronic myeloproliferative neoplasms by droplet digital
PCR ANNALS OF HEMATOLOGY
Waterhouse M, Follo M, Pfeifer D, von Bubnoff N, Duyster J, Bertz H, Finke J
2016. doi:10.1007/s00277-016-2623-0
2015
- Noninvasive monitoring of diffuse large B-cell lymphoma by immunoglobulin high-throughput sequencing
BLOOD
Kurtz DM*, Green MR*, Bratman SV, Scherer F, Liu CL, Kunder CA, Takahashi K, Glover C, Keane C, Kihira S, Visser B, Callahan J, Kong KA, Faham M, Corbelli KS, Miklos D, Advani RH, Levy R, HIcks RJ, Hertzberg M, Ohgami RS, Gandhi MK, Diehn M, Alizadeh AA
2015. doi:10.1182/blood-2015-03-635169
2013
- Detection of mutant free circulating tumor DNA in the plasma of patients with gastrointestinal stromal tumor harboring activating mutations of CKIT or PDGFRA
CLINICAL CANCER RESEARCH
Maier J, Lange T, Kerle I, Specht K, Bruegel M, Wickenhauser C, Jost P, Niederwieser D, Peschel C, Duyster J, von Bubnoff N
2013. doi:10.1158/1078-0432.CCR-13-0765
Unser Labor in den Medien
Wie Krebstherapien von Sportwetten profitieren
27.11.2019 | Mehr
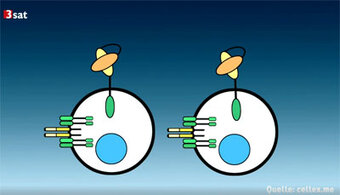
Krebs im Blut aufspüren
Uniklinik Freiburg 07.11.2019 | zum Video
Liquid Biopsy – konzeptionell neuer Ansatz
M&K Kompakt "Labor & Diagnostik" | 11. September 2019 | Mehr
Freiburger Forscher über das Potenzial von Krebserkennungsverfahren
Badische Zeitung | Fr, 22. Februar 2019 | Mehr
Den Krebs im Blut aufspüren
19.10.2017 | "Medizin trifft Technologie" - Vorträge | zum Video
Mit Blut gegen Krebs
20.04.2017 | Besteht ein Verdacht auf Krebs, erfolgt eine Gewebeentnahme, ein invasiver, oft belastender Eingriff. In der Zukunft könnte Patient*innen das erspart bleiben. Gert Scobel diskutiert mit mit seinen... zum Video
[Verfügbar bis 22.04.2022]